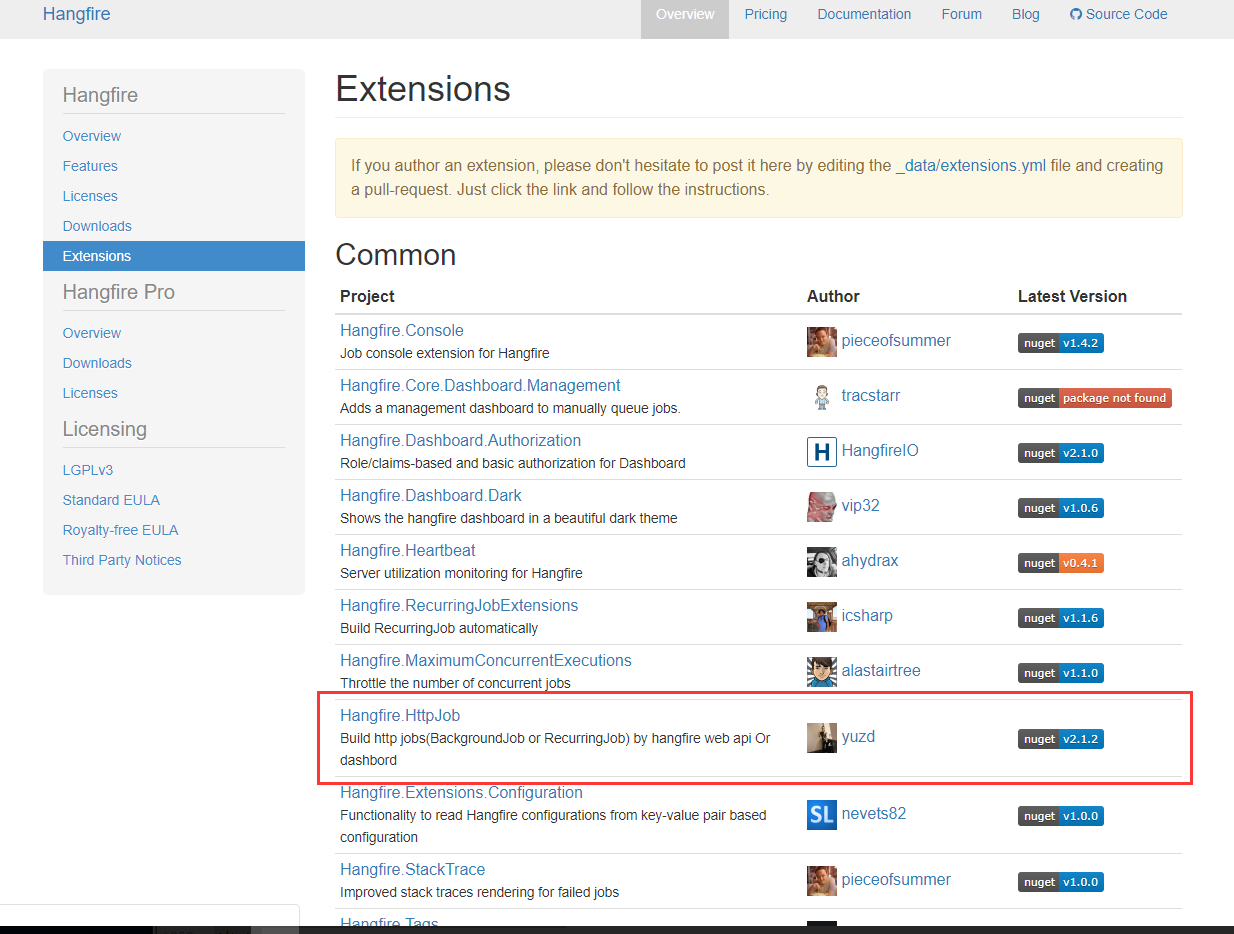
我有一个包含计费数据2001至07年一个数据帧(14.5K行15列)。
我2008年的新数据添加到它: alltime <- rbind(alltime,all2008)
不幸的是,其产生的警告:
> Warning message:
In `[<-.factor`(`*tmp*`, ri, value = c(NA, NA, NA, NA, NA, NA, NA, :
invalid factor level, NAs generated
我的猜测是,有一些新的病人的名字没有在以前的数据帧,因此不知道什么样的水平给那些。 同样新看不见的名字在“转介医生”栏。
有什么解决办法?
Answer 1:
它可以通过类型不匹配两种引起data.frames 。
首先检查类型(类)的。 为了诊断目的这样做:
new2old <- rbind( alltime, all2008 ) # this gives you a warning
old2new <- rbind( all2008, alltime ) # this should be without warning
cbind(
alltime = sapply( alltime, class),
all2008 = sapply( all2008, class),
new2old = sapply( new2old, class),
old2new = sapply( old2new, class)
)
我预计会有一排的样子:
alltime all2008 new2old old2new
... ... ... ... ...
some_column "factor" "numeric" "factor" "character"
... ... ... ... ...
如果是的话解释: rbind不检查类型是否匹配。 如果你分析rbind.data.frame代码,那么你可以看到,第一个参数初始化输出类型。 如果在第一类型data.frame是一个因素,然后输出data.frame列因子与水平unique(c(levels(x1),levels(x2))) 但是当第二data.frame列不那么因子levels(x2)为NULL ,所以水平不延长。
这意味着,你的输出数据是错误的! 有NA的,而不是真正的价值
我想:
- 你创建你的旧数据与另一R / RODBC版本,所以类型用不同的方法创建的(不同的设置 - 小数点分隔也许)
- 有空的或有问题的塔,例如一些特定的数据。 有人在数据库中更改列。
解:
找错列,并找出原因,它的错误的和固定的。 消除病因尚未症状。
Answer 2:
“简单”的方法是根本就没有导入文本数据时,有你的字符串设定为因素。
需要注意的是read.{table,csv,...}功能采取stringsAsFactors参数,这是默认设置为TRUE 。 你可以将其设置为FALSE ,当你导入和rbind -ing您的数据。
如果您想设置列在年底的一个因素,你可以做到这一点。
例如:
alltime <- read.table("alltime.txt", stringsAsFactors=FALSE)
all2008 <- read.table("all2008.txt", stringsAsFactors=FALSE)
alltime <- rbind(alltime, all2008)
# If you want the doctor column to be a factor, make it so:
alltime$doctor <- as.factor(alltime$doctor)
Answer 3:
1)创建数据帧与stringsAsFactor设置为FALSE。 这应该可以解决因子的问题
2)之后不使用rbind - 它搅乱了列名,如果该数据帧是空的。 简单地做这种方式:
df[nrow(df)+1,] <- c("d","gsgsgd",4)
/
> df <- data.frame(a = character(0), b=character(0), c=numeric(0))
> df[nrow(df)+1,] <- c("d","gsgsgd",4)
Warnmeldungen:
1: In `[<-.factor`(`*tmp*`, iseq, value = "d") :
invalid factor level, NAs generated
2: In `[<-.factor`(`*tmp*`, iseq, value = "gsgsgd") :
invalid factor level, NAs generated
> df <- data.frame(a = character(0), b=character(0), c=numeric(0), stringsAsFactors=F)
> df[nrow(df)+1,] <- c("d","gsgsgd",4)
> df
a b c
1 d gsgsgd 4
Answer 4:
如前面的回答表明,阅读列,字符和后做转换的因素rbind 。 SQLFetch (I假设RODBC)还具有stringsAsFactors或as.is参数,以控制字符的转换。 允许的值作为read.table ,例如as.is=TRUE或一些列号。
Answer 5:
我曾与类型不匹配了同样的问题,尤其是与因素。 我不得不胶水两个原本兼容的数据集中在一起。
我的解决办法是因素都dataframes转换为“性格”。 然后,它就像一个魅力:-)
convert.factors.to.strings.in.dataframe <- function(dataframe)
{
class.data <- sapply(dataframe, class)
factor.vars <- class.data[class.data == "factor"]
for (colname in names(factor.vars))
{
dataframe[,colname] <- as.character(dataframe[,colname])
}
return (dataframe)
}
如果你想看到你的两个dataframes类型运行(其他城市VAR名):
cbind("orig"=sapply(allSurveyData, class),
"merge" = sapply(curSurveyDataMerge, class),
"eq"=sapply(allSurveyData, class) == sapply(curSurveyDataMerge, class)
)
Answer 6:
当您创建数据帧你让你的字符串列因素(的选择stringsAsFactors=T ),或让他们为字符串。
对于你的情况下,不要让你的字符串列因素。 让他们为字符串,然后追加工作正常。 如果你需要他们最终会因素,做所有的插入和第一追加为字符串,那么最后将它们转换为因素。
如果您的字符串列因素,然后添加含有看不见的值的行,你得到你的每一个新的看不见的因素水平提到的错误和价值获取与NA取代...
> df <- data.frame(patient=c('Ann','Bob','Carol'), referring_doctor=c('X','Y','X'), stringsAsFactors=T)
patient referring_doctor
1 Ann X
2 Bob Y
3 Carol X
> df <- rbind(df, c('Denise','Z'))
Warning messages:
1: In `[<-.factor`(`*tmp*`, ri, value = "Denise") :
invalid factor level, NA generated
2: In `[<-.factor`(`*tmp*`, ri, value = "Z") :
invalid factor level, NA generated
> df
patient referring_doctor
1 Ann X
2 Bob Y
3 Carol X
4 <NA> <NA>
所以, 不要让你的字符串列因素。 让他们为字符串,然后追加正常工作 :
> df <- data.frame(patient=c('Ann','Bob','Carol'), referring_doctor=c('X','Y','X'), stringsAsFactors=F)
> df <- rbind(df, c('Denise','Z'))
patient referring_doctor
1 Ann X
2 Bob Y
3 Carol X
4 Denise Z
要更改默认的行为 :
options(stringsAsFactors=F)
各个列转换为/从字符串或因素
df$col <- as.character(df$col)
df$col <- as.factor(df$col)
Answer 7:
这里取2个数据帧的共同行名字,做一个rbind我们基本上发现,一些因素的字段,添加新的因素,然后做rbind的功能。 这应该照顾的任何因素的问题:
rbindCommonCols <-function(X,Y){
commonColNames = intersect(colnames(x), colnames(y))
x = x[,commonColNames]
y = y[,commonColNames]
colClassesX = sapply(x, class)
colClassesY = sapply(y, class)
classMatch = paste( colClassesX, colClassesY, sep = "-" )
factorColIdx = grep("factor", classMatch)
for(n in factorColIdx){
x[,n] = as.factor(x[,n])
y[,n] = as.factor(y[,n])
}
for(n in factorColIdx){
x[,n] = factor(x[,n], levels = unique(c( levels(x[,n]), levels(y[,n]) )))
y[,n] = factor(y[,n], levels = unique(c( levels(y[,n]), levels(x[,n]) )))
}
res = rbind(x,y)
res
}
文章来源: Unseen factor levels when appending new records with unseen string values to a dataframe, cause Warning and result in NA